기사본문
"동반진단, 액체생검..." 확산되는 국내 암 유전체 연구
입력 2017-02-22 15:11 수정 2017-02-22 15:11
김태형 테라젠이텍스 이사
지난 8~10일 강원도 홍천 비발디파크에서 한국유전체학회 동계심포지엄이 열렸다. 김태형 테라젠이텍스 이사가 이번 심포지엄의 주요 발표 내용을 정리해 보내왔는데 국내외 유전체 연구 트렌드를 살펴볼 수 있어 소개한다. -편집자주
질병을 맞춤진단하고 맞춤치료하는 정밀의료 시대로 패러다임이 바뀌는 시점에서 이번 한국유전체학회 심포지엄은 매우 뜻 깊었다. 최근 급속히 발전하고 있는 유전체 분석 기술과 이 기술을 이용한 국내 연구자들의 다양한 유전체 연구를 살펴볼 수 있는 자리였기 때문이다. 특히 국내 암 유전체 연구의 발전상을 엿볼 수 있었다.
먼저 김권일 카이스트 박사는 작년 말 네이처 제네틱스(Nautre Genetics)에 “Chromatin structure–based prediction of recurrent noncoding mutations in cancer”라는 제목으로 논문을 출판한 머신러닝(Machine learning)을 활용한 암 게놈 빅데이터 연구를 소개했다.
이 연구는 단백질 정보를 담고 있지 않은 Junk DNA에서 발생하는 돌연변이가 복잡한 3차원 DNA 구조를 통해 단백질 정보를 담고 있는 부분과 상호작용함으로써 암의 유발 인자가 될 수 있음을 밝힌 것으로 연구팀은 암 게놈 빅데이터를 통해 junk DNA 돌연변이를 예측하는 방법을 개발했다.
연구팀은 유방암과 폐암 샘플에서 3차원 크로마틴 상호작용에 근거해 재발하는 체세포 변이(somatic mutation)를 측정해 기존에 발견하지 못한 체세포 변이 및 암 관련 유전자를 새롭게 찾아내는 추론 모델을 구축했다. 이후 암환자 코호트 게놈 빅데이터와 머신러닝 기술을 이용해 단백질 비번역(noncoding) 영역에서의 암을 유발하는 새로운 암 유발 돌연변이(driver mutation)를 예측했다.
김 박사의 발표는 국내에서 생산되는 제한된 게놈 데이터와 임상 데이터를 활용해 좀 더 가치 있는 데이터로 전환할 수 있는 머신러닝의 활용 가치를 국내 연구자들에게 전달한 것이다.
우현구 아주의대 교수는 약물유전체 데이터 분석을 통한 항암제의 신규 타겟을 발굴하는 연구결과를 소개했다. 우 교수는 CCLE(Cancer Cell Line Encyclopedia)와 GDSC(Genomics of Drug Sensitivity in Cancer)를 활용해 14개의 후보 약물-변이의 연관성을 예측하고 발굴해 실제 간암 환자에게 잘 반응을 하는 TKI, VEGF 저해제로 잘 알려진 넥사바(Nexavar, 성분명:Sorafenib)를 처방했을때 SULF2(Sulfatase 2) 유전자 변이와 발현 차이를 어떻게 보이는지 아주대 병원과 세브란스 병원의 간암 환자 코흐트를 이용해 확인했다. 그 결과 SULF2 뿐만 아니라 LCN2 유전자도 발견해 간암 환자에 넥사바를 처방할때 이들 유전자에 대해서는 동반진단에 적용될 가능성이 보인다고 발표했다.
이승태 세브란스병원 교수(진단검사의학과)는 세브란스병원에서 준비하는 “NGS Panels” 소개했다. 자폐증부터 피부질환 및 혈액암 패널까지 자체 디자인해 제작한 20여가지 NGS 유전자 패널에 대한 것이었다. 올해 3월 1일부터 암 환자를 대상으로 수십, 수백 개 암 유전자를 스크리닝하는 암유전체 검사에 대해 50% 보험급여가 적용되는 상황에서 의미있는 발표였다.
이번 학회에서는 액체생검(ctDNA) 관련된 발표들도 많았다. 액체생검은 진단시장의 퀀텀점프를 가져올 혁신적인 기술로 전세계적으로 활발한 연구가 진행되고 있다.
김지연 삼성서울병원 교수(혈액종양내과)는 국소 진행성 유방암 (Locally advanced breast cancer) 환자들을 대상으로 선행화학요법(Neoadjuvant chemotherapy)을 적용하면서 효과적인 시리얼 모니터링을 통해 ctDNA(Circulating Tumor DNA)를 표지자로 적용한 사례를 발표했다.
혈장(Plasma) DNA와 암조직 DNA를 동시에 얻어 ctDNA를 모니터링하고 이를 “흉부 CT”와 “유방 자기공명영상(Breast MRI)” 자료와 비교하는 방식으로 연구를 진행했는데 대부분 환자에서 TP53과 BRCA1/2에서 변이가 많이 발견된 것을 확인할 수 있었다. 결국 임상 결과와 ctDNA의 양적 변화는 관련성이 매우 높았고 “장기추적관찰(long term followup)”의 경우 환자 치료에 도움이 됐다고 설명했다.
이번 학회에서는 세계 최초 FDA에 허가받은 ctDNA기술인 Cobas와 NGS 기반의 액체생검 기술을 이용한 동반진단(폐암) 기술도 소개됐다. 삼성유전체연구소(SGI)의 박동현 박사는 ctDNA 기술을 이용해 종양 부하(tumor burden)를 모니터링한 결과를 소개했고 바코딩 기술을 추후 적용해 중복률(duplication rate)를 현격히 줄이고 이를 통해 발생한 에러를 줄여나가는 방향으로 기술을 업그레이드하겠다고 발표했다.
또한 ICGC(International Cancer Genome Consortium, 국제암유전체컨소시엄)가 거의 마무리되는 시점에서 한국에서도 이 게놈 데이터를 분석한 발표도 많았다. ICGC는 임상적·사회적 중요성이 큰 50여종의 암에 대해 유전체, 전사체, 후성유전체 등의 국제 공동연구를 수행하는 컨소시엄이다.
서울대학교병원 고영일 교수(혈액종양내과)는 KIR(killer-cell immunoglobulin-like receptor) 유전자 들은 HLA 유전자들과 마찬가지로 제노타이핑 하기에 매우 어려운 영역인데 국제암유전체 컨소시엄(International Cancer Genome Consortium,ICGC)의 수천 개의 WES/WGS 데이터를 이용해 성공적으로 수행한 경험을 소개했다.
특정 합플로타입(Haplotype)을 가진 간암, 전립선암, 췌장암, 위암 그리고 직장암 환자들에서는 암 발병률이 높아지는 것을 볼 수가 있었다. 특히 HBV에 의한 간암 환자와 관련성을 보였다. 그리고 편평세포암종(squamous cell carcinoma)에서는 예후가 매우 안 좋았다.
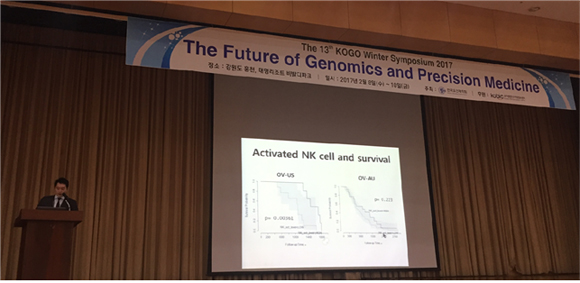
KIR-HLA에 대한 복잡하고 어려운 분석을 통해 “NK cell”의 활성에 대한 단서를 발견했는데 NK cell 불활성화 현상(inacvation)은 만성림프구성 백혈병(chronic lymphocytic leukemia, CLL), 피부암(Melanoma) 그리고 두경부 편평세포암(Head and Neck Squamous Cell Carcinoma, HNSCC)에서 발생했다. 특히 난소암에서 “NK cell”의 활성도는 생존율과 매우 관련성이 높았다. 고 교수는 KIR-HLA의 제노타입을 예측하고 스코링 함으로써 암 환자에서 “NK cell”의 활성도를 측정할 수 있을 것으로 예측했다.
카이스트 주영석 교수는 ICGC 데이터 중 암 조직 21개 타입에서 암 환자 2658명의 암/정상 게놈 데이터 16.5kb 사이즈의 mtDNA를 분석해 최종적으로 2,536명의 샘플에서 7,611개 mtDNA 체세포 변이치환(somatic substitution)을 발견했다. 주 교수는 특정 mtDNA 영역에서 짧은 시간 안에 과돌연변이가 일어나는 것을 발견했는데 재밌는 것은 mtDNA 카피수가 조직마다 차이가 나는데 적게는 수십 개에서 많게는 1,000여 개 까지 다양하게 존재한다. 암 조직에서는 평균 200-300 mtDNA 카피를 발견됐는데 그중에서 자궁경부에서 가장 적게 존재하고 난소와 간에서 가장 mtDNA 카피 수가 많았다. 또한 mtDNA에서 C>T, T>C 돌연변이가 많이 발견되었는데 이는 담배나 자외선 같은 발암물질(carcinogen)과 같은 외부적 요인이 아니라 복제중에 생기는 자연 발생적 변이로 보인다.
서울대병원 윤성수 교수(혈액종양내과)는 ICGC(2008년~2018년) 후속으로 진행되는 ICGCmed(2016년~2025년)에 대해 소개했다. ICGCmed는 2016년 4월 18일 AACR16 컨퍼런스에서 공식적으로는 발표했으며 환자의 게놈과 임상 및 헬스 정보를 결합해서 연구하는 것으로 확장하는 프로젝트이다.
최종 목표는 기존 프로젝트보다 4배나 규모가 큰 2016년~2025년까지 약 10년 동안 임상 정보가 있는 암 환자 20만 명의 게놈을 확보하는 것을 목표로 하고 있다. 그는 "ICGCmed는 WES, WGS, cfDNA 및 immune-marker(면역 지표) 검사등의 검사를 통해 암 유발 돌연변이(driver alteration)을 검출하는 비율을 90%까지 끌어 올리고 최종적으로 100명의 암 환자 중에 60명 이상에서 타깃 항암제가 환자에게 이득을 보게 하기 위한 프로젝트"라고 소개했다.
전 세계적으로도 암유전체 연구가 실질적인 암환자 치료에 적용함에 있어 큰 어려움이 있었지만, 이를 해결하는 방법들을 찾아 나아가는 것을 보면서 국내에서도 내년 한국 유전체학회 동계심포지엄에서는 실질적으로 환자가 혜택을 보는 성공 케이스들을 선보이지 않을까 하는 기대를 가져 본다.







